 |
 |
 |
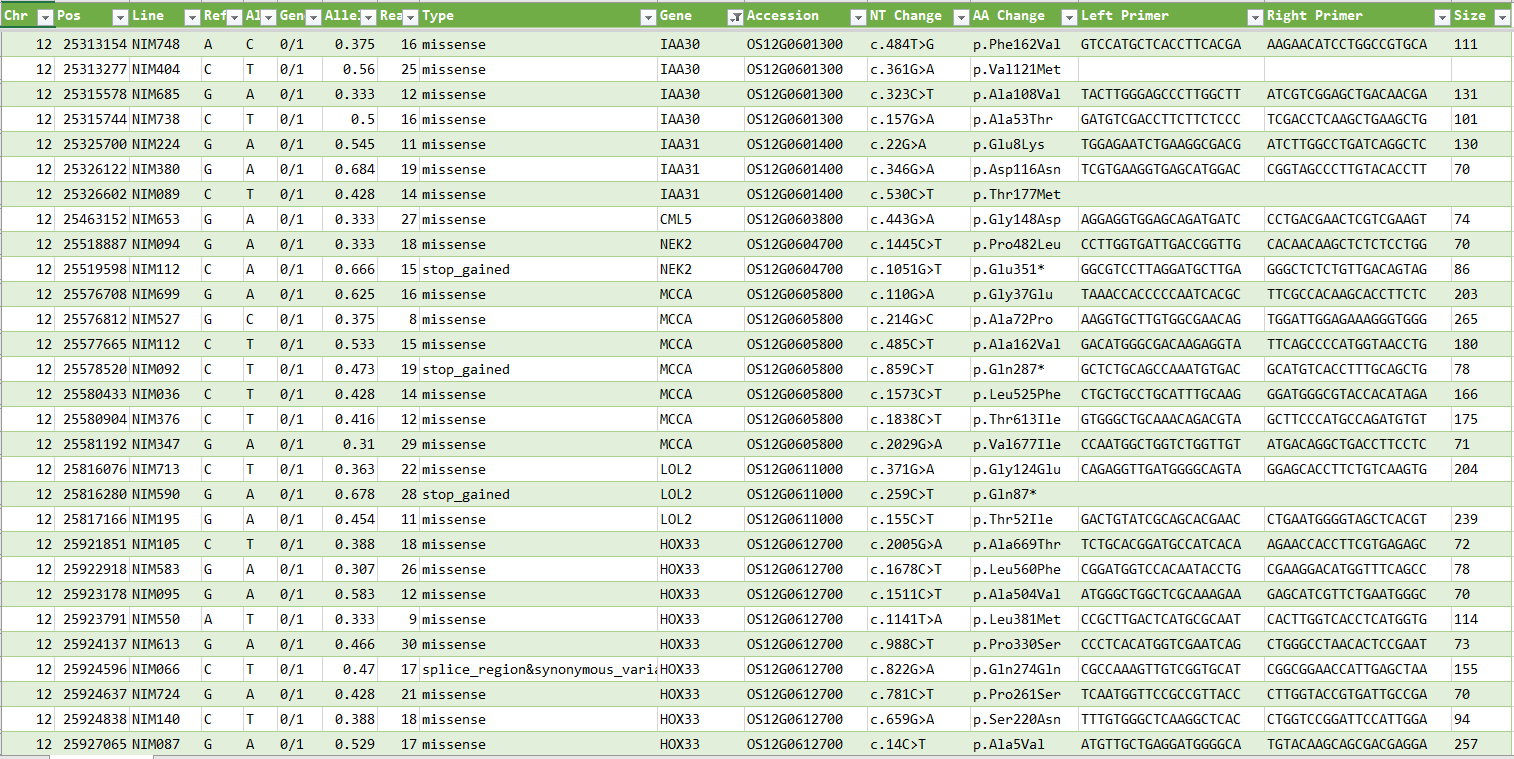
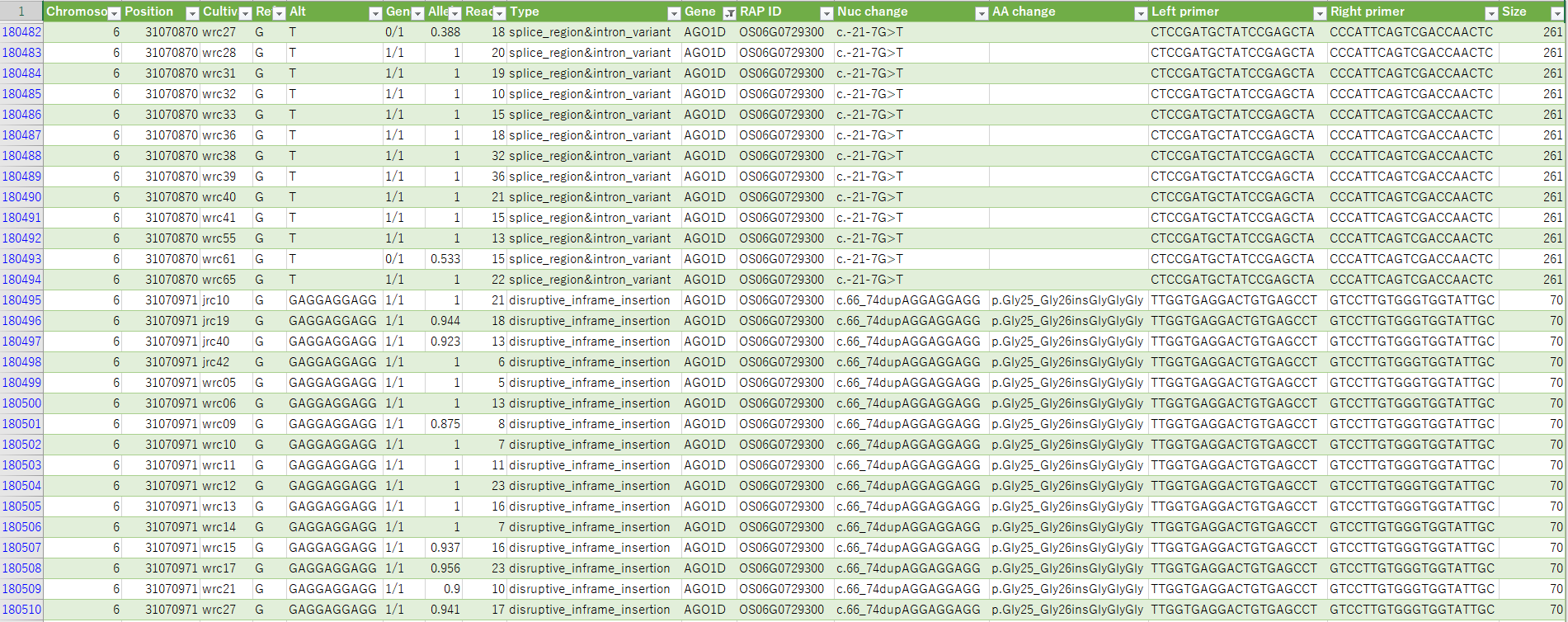
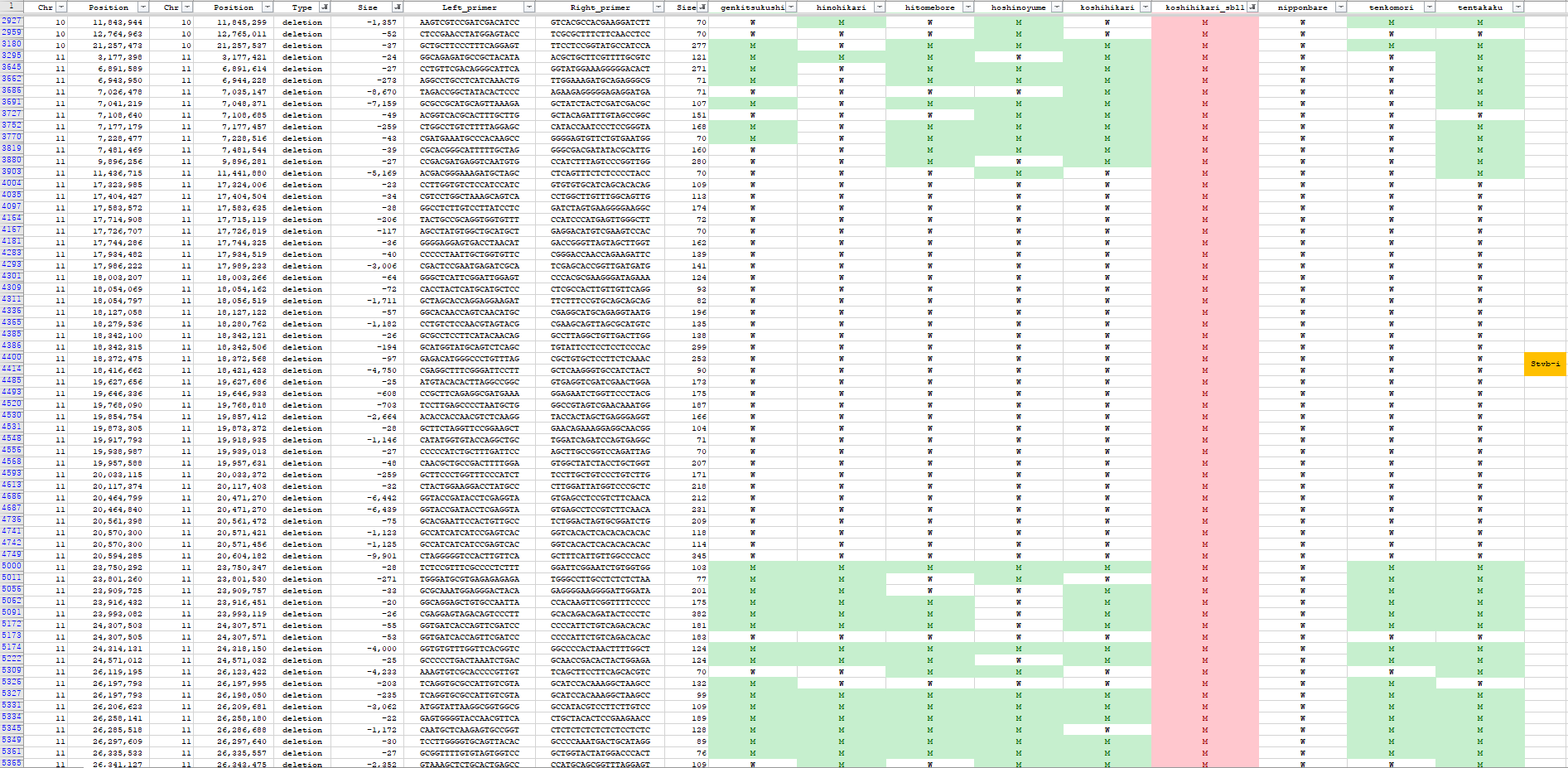
| 日本晴MNU変異系統のSNP一覧 New | イネコアコレクションの多型一覧 | コシヒカリSBL1のSBL1特異的領域の検出 |
 |
 |
 |
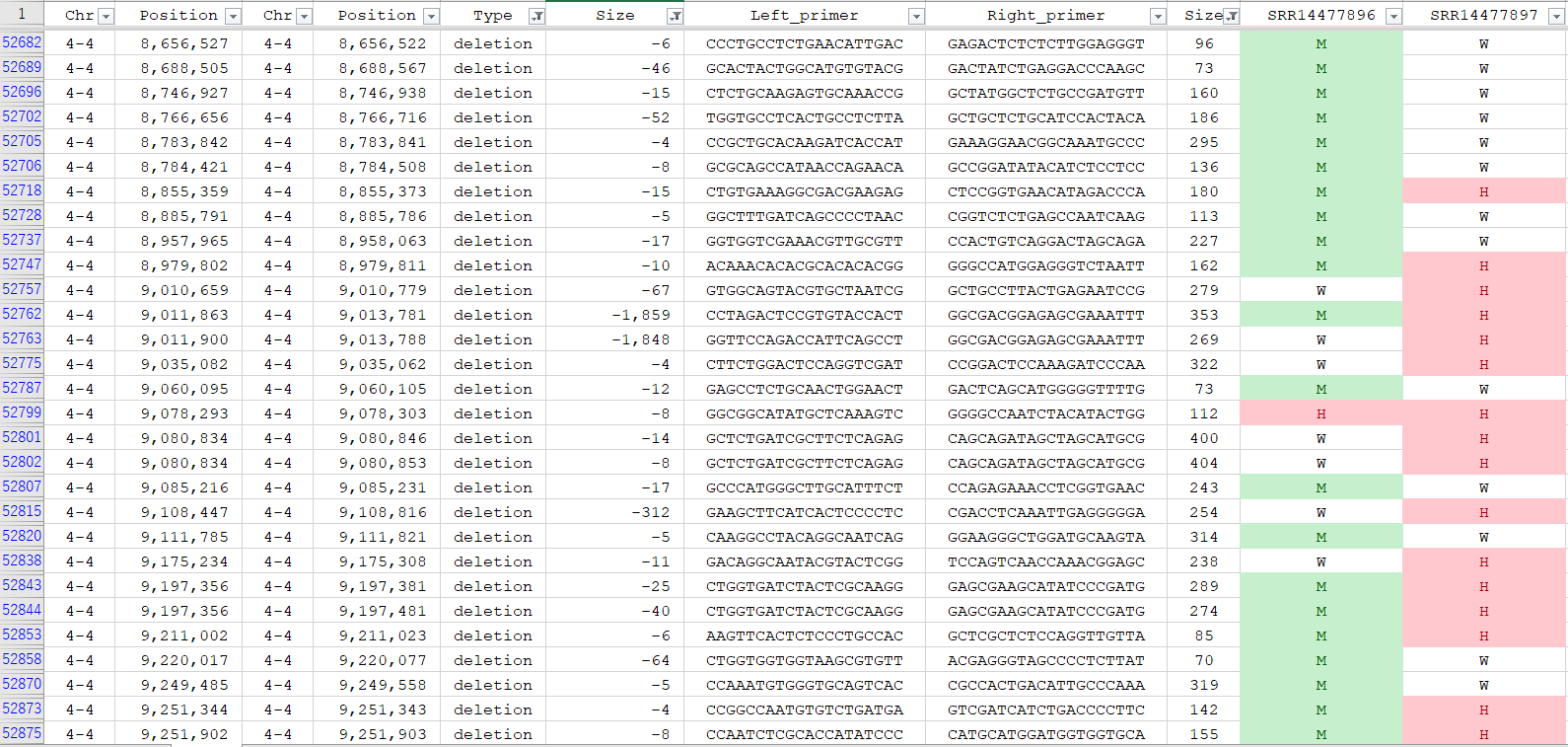
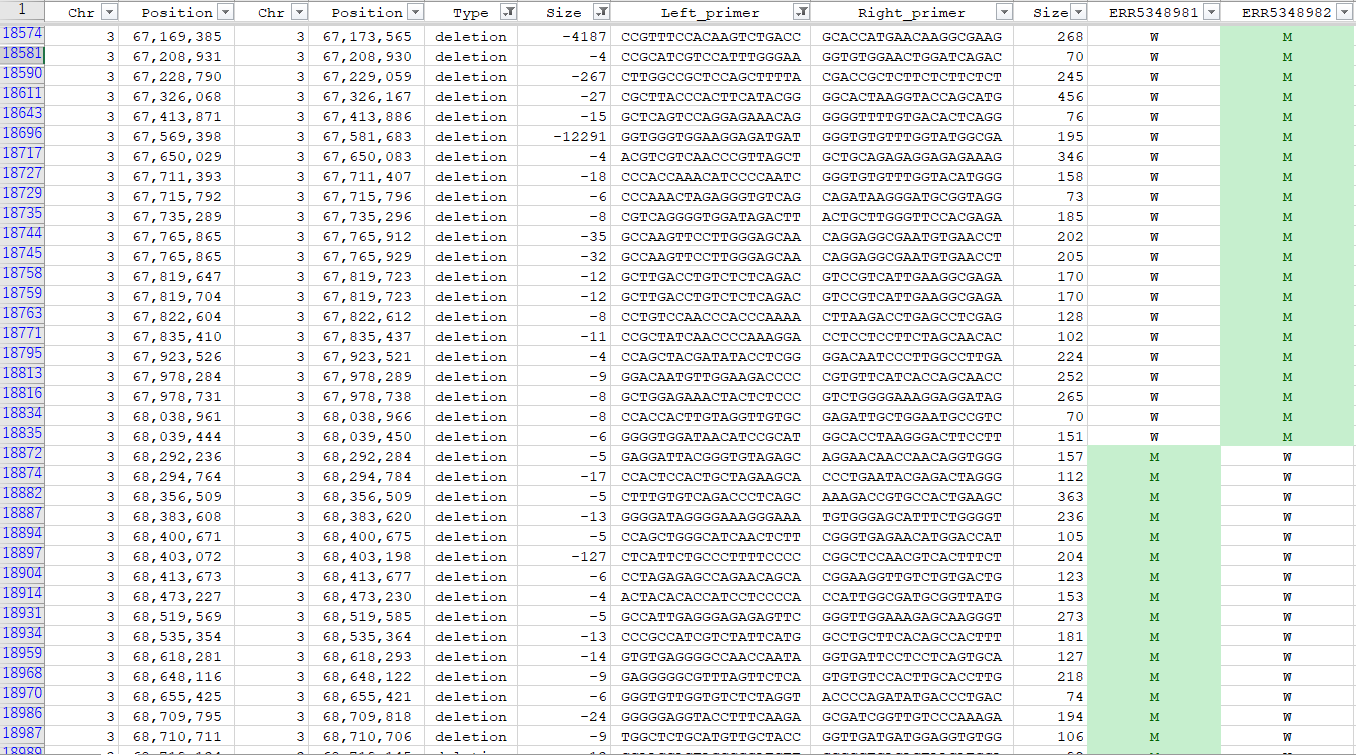
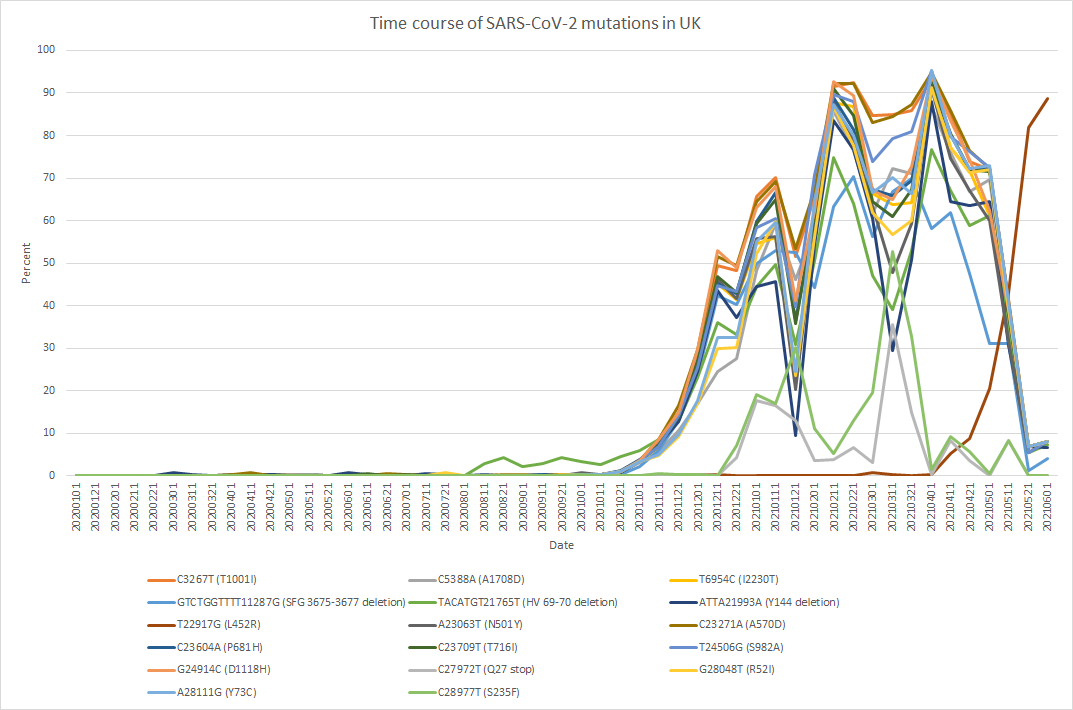
| 異質8倍体のイチゴの構造変異の検出 | トマトの構造変異の検出 | 1ヶ月でL452R変異を持つデルタ株と入れ替わりました。 |
Polymorphic Edge Detection (PED)は、次世代シーケンサーを用いて得られた塩基配列(ショートリード)より、配列の多型のキワ(エッジ)に着目して効率よく多型を検出する方法です。
非常に正確にゲノム全体に渡るSNPとindel等構造変異を得ることができます。
次世代シーケンサーの配列を解析する場合、まず配列をリファレンスゲノム配列にマップして、不一致の部分を抽出するのが一般的ですが、大部分のショートリードは多型を含まないため効率はあまりよくありません。
PEDでは、ショートリードから多型のエッジになる部分を早い段階で抽出するアルゴリズムで、これまでにない新しい手法です。
エッジを検出する方法として、ショートリードを長さk(k=20)の細かい断片に分解してターゲットとコントロールのショートリードを直接比較して多型を検出する方法(k-mer法)と、ショートリードに対してリファレンスゲノム配列を5'側からと3'側から双方向に整列してエッジを検出する双方向整列法(Bidirectional Alignment法)の2つのアルゴリズムを実装しています。
k-mer法はリファレンス配列がなくても、3'末端に多型を持つk-merを抽出することができ、連鎖解析に利用できます。
双方向整列法は、SNPに加えて、挿入、欠失、逆位、転座などの構造変異が同時に検出されます。また、従来法に比べて正確かつ高速に処理することができ、特に数100kbを超えるような大きな欠失も正確に検出できる特徴があります。
シンプルなアルゴリズムなので、ヒトの5倍の大きさで6倍体ゲノムのコムギのような複雑なゲノム構成の生物の多型も検出できます。また、小規模なコンピュータリソースで動作が可能です。 書式
ped.plでの多型検出
時間はかかりますが、シングルコアや2コアのCPUでも動作します。MacOSでも動作しますが遅いです。
メモリーは4Gバイト以上、ディスクはバクテリアなら数Gバイト、ヒトを解析する場合はディスクは1TB程度必要です。
コードはGithubから、実行に必要なファイルが設定ずみのDockerのコンテナも公開しています。AWSやGCP等のクラウド上での実行も可能です。
perl ped.pl target=target,ref=reference
perl ped.pl target=target,control=control,ref=reference
$ sudo apt update
$ sudo apt upgrade
$ sudo apt install curl
$ sudo apt install git
$ sudo apt install sra-toolkit
$ git clone https://github.com/akiomiyao/ped.git
cd ped
mkdir your_sample1
mkdir your_sample1/read
cp somewhere/your_shortread.fastq your_sample1/read
perl ped.pl target=your_sample1,ref=reference_name
perl ped.pl
と引数なしで実行すると、サポートしているリファレンスが一覧されます。
その中のリファレンス名を指定してください。
サポートされていないリファレンスで解析したい場合は、実行例の一番下をご参照ください。
targetの名前を決めて、決めたtargetの名称のディレクトリを作り、その中にreadという名称のディレクトリを作って、readディレクトリの中にfastqファイルを置きます。
fastqファイルは、.gz、.bzip2、.xzなどの拡張子のついた圧縮ファイルでも大丈夫です。
readディレクトリ内のファイルの圧縮形式は1種類のみにしてください。
targetのディレクトリの中に、結果がセーブされます。
NCBIで公開されている配列データを解析する場合は、
perl download.pl accession=Accesion_name
で、Accession_nameで指定したディレクトリができて、その中のreadディレクトリにfastqファイルがダウンロードされます。
ヒトのhg38のリファレンスに対して、イルミナのプラチナゲノム配列の一つERR194146(CEPH/UTAH家系の父親)の多型を検出します。
Dockerのコンテナを用いて、イネのIRGSP1.0のリファレンスに対するコシヒカリ(DRR054198)の多型を検出します。Dockerの設定は、こちら
NCBIのSRAアーカイブよりアクセッション番号SRR11513115のfastqファイルをダウンロードします。
SRR11513115/read のディレクトリにfastqファイルが納められます。
長さが不揃いなRT-PCRの配列データを146塩基にそろえて、新型コロナウイルスのリファレンス配列に対する多型を検出します。
結果はSRR11513115のディレクトリに納められます。
最新のped.plでは、長さの不揃いな配列データから最適リード長を算出して自動で切りそろえるようになりました。clippingオプションは原則不要です。
自動認識のclipping長とは別の長さで解析したい場合のみclippingオプションを指定してください。
長さが不揃いな配列データの各リード長に対するリード数を出力します。最大リード長より少し短めにしてclippingの値とすると効率がよくなります。
線虫の継代サンプルERR3063487と継代元サンプルERR3063486で継代によって発生した自然突然変異の検出の場合。
ショートリードデータやリファレンスデータの入ったディレクトリを置く作業ディレクトリを/home/your_id/dataに指定する場合です。
指定しない場合は、ped.plがあるディレクトリが作業ディレクトリになります。
最大スレッド(プロセス)数を指定する場合。通常は設定する必要はありませんが、同じコンピュータで同時に別の作業もする場合は、プロセスの上限を下げると影響が少なくなります。
現在ではthreadによる分散処理からprocessベースに移行しています。オプション名は、正確にはprocessとすべきですが、混乱をさけるためthreadのままにしてあります。
一時ファイル置き場に高速ディスクを指定する場合。
k-mer法で解析する場合。デフォルトは双方向整列法。
リファレンスのみを指定すると、リファレンスだけが作られます。
mkdir corn
cp ref_maize.fasta.gz corn
perl ped.pl ref=corn,file=ref_maize.fasta.gz
cornというディレクトリに置いたfastaファイル(圧縮可)からリファレンス用データセットができます。
ped.plでの解析では、ref=cornと指定します。
大きな構造変異を調べたい場合は、targetのディレクトリの中に出力された、svの拡張子のついたファイルを直接ご参照ください。
詳しくは、ページの一番最初にある「解析例のショートカット」をご参照ください。
pedのディレクトリ内で、
perl download.pl accession=ERR3063486
perl download.pl accession=ERR3063487
を実行します。(アメリカからのダウンロードなので、数時間程度かかります。fastq-dumpが必要です。インストール法はこちら)
dockerで実行する場合は、
docker run -v `pwd`:/work -w /ped akiomiyao/ped perl download.pl accession=ERR3063486,wd=/work
docker run -v `pwd`:/work -w /ped akiomiyao/ped perl download.pl accession=ERR3063487,wd=/work
を実行します。
このデモでは、線虫のコントロール(ERR3063486)に対して、250世代後の線虫(ERR3063487)に生じた自然突然変異をWBcel235のリファレンスゲノムを用いてbidirctional法で検出しています。
pedのディレクトリ内で、
perl ped.pl target=ERR3063487,control=ERR3063486,ref=WBcel235
あるいは、
docker run -v `pwd`:/work -w /ped akiomiyao/ped perl ped.pl target=ERR3063487,control=ERR3063486,ref=WBcel235,wd=/work
とすると、現在のカレントディレクトリ上に存在するERR3063486、ERR3063487、WBcel235のディレクトリ内のファイルが解析対象になります。
結果は、ターゲットのERR3063487のディレクトリに結果が出力されます。
targetの部分はターゲットとして指定したディレクトリ名となります。
target.vcf HとMでマークされた多型をvcf形式で出力されたファイル。IGVで表示させるために転座や大きな欠失は除いている target.full.vcf HとMでマークされた多型をvcf形式で出力されたファイル。大きな欠失等を除く前のデータ target.count.vcf HとMでマークする前の、target.alnで検出された多型の検出回数で整理し、vcf形式で出力されたファイル target.aln 双方向整列法による整列データ target.bi.snp 双方向整列法によるSNPリスト 確度の高いSNPはH(ヘテロ)とM(変異型ホモ)でマークされます target.bi.primer 双方向整列法によるSNPリストに加えてプライマー配列が出力されます target.kmer.snp k-mer法で検出されたSNPリスト target.kmer.primer k-mer法で検出されたSNPリストに加えてプライマー配列が出力されます target.sv 双方向整列法で検出された構造(挿入・欠失・逆位・転座)変異リスト target.sv.primer 双方向整列法で検出された構造(挿入・欠失・逆位・転座)変異リストに加えてプライマー配列が出力されます target.index アラインメント検索用インデックスファイル
perl search.pl target=target,chr=chromosome,pos=position
を実行します。
例
perl search.pl target=ERR3063487,chr=II,pos=948033
Dockerコマンドで実行する場合は、
docker run -v `pwd`:/work -w /ped -t akiomiyao/ped perl search.pl target=ERR3063487,chr=II,pos=948033,wd=/work
vcfファイルでは挿入・欠失の開始位置が、多型の一つ前の塩基となっています。
挿入・欠失のアラインメントをsearch.plで検索する場合は、posの値はvcf形式の結果ファイルの位置に1を加えた値にしてください。
結果のフォーマット
ERR194146のディレクトリの中にERR194146.vcf
というvcf形式マップデータがセーブされます。
vcf形式の結果はBroad Instituteから提供されているIntegrative Genomics Viewer (IGV)でグラフィカルに表示可能です。
##fileformat=VCFv4.2
##INFO=<ID=AF,Number=.,Type=Float,Description="Allele Frequency">
##INFO=<ID=DP,Number=1,Type=Integer,Description="Approximate read depth)">
##INFO=<ID=END,Number=1,Type=Integer,Description="End position of the variant described in this record. Edge position of the alignment from 3'-end of short read is shown as END.">
##INFO=<ID=GT,Number=1,Type=String,Description="Genotype">
##INFO=<ID=HOMLEN,Number=.,Type=Integer,Description="Length of base pair identical micro-homology at event breakpoints">
##INFO=<ID=HOMSEQ,Number=.,Type=String,Description="Sequence of base pair identical micro-homology at event breakpoints">
##INFO=<ID=PCRLEN,Number=.,Type=Integer,Description="PCR product length">
##INFO=<ID=PL,Number=.,Type=String,Description="Left primer sequence">
##INFO=<ID=PR,Number=.,Type=String,Description="Right primer sequence">
##INFO=<ID=SVLEN,Number=.,Type=Float,Description="Difference in length between REF and ALT alleles">
##INFO=<ID=SVTYPE,Number=1,Type=Integer,Description="Type of structural variant">
##ALT=<ID=DEL,Description="Deletion">
##ALT=<ID=INS,Description="Insertion of novel sequence">
##ALT=<ID=INV,Description="Inversion of reference sequence">
##FORMAT=<ID=GT,Number=1,Type=String,Description="Genotype">
##FORMAT=<ID=AD,Number=.,Type=Integer,Description="Allelic depths for the reference and alternate alleles in the order listed">
##FORMAT=<ID=DP,Number=1,Type=Integer,Description="Read depth">
##source=<PROGRAM=ped.pl,Method="Bidirectional method",target=ERR194146,control=hg19,reference=hg19>
##created=<TIMESTAMP="2020-05-07 19:45:32 +0900">
#CHROM POS ID REF ALT QUAL FILTER INFO FORMAT ERR194146
1 69511 . A G 1000 . GT=1/1;AF=1;DP=19;PL=TGTGTGGCAACGCATGTGTC;PR=CAACTGGCTCACCGAATGGA;PCRLEN=74 GT:AD:DP 1/1:0,19:19
1 564654 . G A 1000 . GT=0/1;AF=0.534;DP=43;PL=CTCTCCCCTGAACTCTACAC;PR=CGGGGGTATGCTGTTCGAAT;PCRLEN=89 GT:AD:DP 1/1:20,23:43
1 564655 . A C 1000 . GT=0/1;AF=0.343;DP=32;PL=CTCTCCCCTGAACTCTACAC;PR=CGGGGGTATGCTGTTCGAAT;PCRLEN=89 GT:AD:DP 1/1:21,11:32
1 564898 . A C 1000 . GT=0/1;AF=0.313;DP=67;PL=TCCAGCATTCCCCCTCAAAC;PR=GGTGGCACGGAGAATTTTGG;PCRLEN=144 GT:AD:DP 1/1:46,21:67
1 565006 . T C 1000 . GT=0/1;AF=0.666;DP=57;PL=TAAGCTATCGGGCCCATACC;PR=GGTTGGGCCAGGGGATTAAT;PCRLEN=72 GT:AD:DP 1/1:19,38:57
1 565006 . T G 1000 . GT=0/1;AF=0.666;DP=57;PL=TAAGCTATCGGGCCCATACC;PR=GGTTGGGCCAGGGGATTAAT;PCRLEN=72 GT:AD:DP 1/1:19,38:57
1 565454 . T C 1000 . GT=0/1;AF=0.347;DP=23;PL=CCTGCTCCTTCTCACATGAC;PR=GAGTGAGGAGAAGGCTTACG;PCRLEN=89 GT:AD:DP 1/1:15,8:23
1 565464 . T C 1000 . GT=0/1;AF=0.514;DP=35;PL=CCTGCTCCTTCTCACATGAC;PR=GAGTGAGGAGAAGGCTTACG;PCRLEN=89 GT:AD:DP 1/1:17,18:35
1 833173 . C CGAAC 1000 . SVTYPE=INS;END=833173;SVLEN=4;PL=GCTTGCTGAATTCCTGCCTG;PR=GAGGACTGTGGGCGAAAAGT;PCRLEN=73;GT=0/1;AF=0.473;DP=38 GT:AD:DP 0/1:20,18:38
1 867997 . CTTTC C 1000 . SVTYPE=DEL;END=867998;HOMLEN=4;HOMSEQ=TTTC;SVLEN=-4;PL=CTCCCAGACCATGTCTGTGT;PR=AACACACAGATGGAGGTGGC;PCRLEN=85;GT=1/1;AF=0.969;DP=33 GT:AD:DP 1/1:1,32:33
PEDでは、bwaを使わず独自の方法でマッピングしているため、bwa/GATKと同様のクオリティースコアの算出が困難です。
そのため、クオリティースコアは1000の固定値としています。
多型のクオリティーは、サポートされるリードのカバー数(DP)で判断して下さい。
転座に関してはIntegrative Genome Veiwer(IGV)でうまく表示できないので含めていません。同時に出力されるsvのファイルをご参照ください。
1 1223251 A G 50 0 0 26 M GCTGAGCACTCACTTTCCTC ATAGCGAGAAGGAGGTTGCC 76 CACACGGCACGCACACACGTACTCACGAGCACACGCACGGCACACTCATACACGACACACACGGCACACTCATGTACACACAACACACGCGCGCACACACAGGATGCCTGAGACCGGTGGCGGGAGCCTGGCTGAGCACTCACTTTCCTC[G/A]TCCACTTTGAGTTCCTCGTTGAGCCTGATGGCGTCGGCAACCTCCTTCTCGCTATGGCCTTTACTCGCCAAAAACTTCACCTTATACTCGTCCCAAGACACGTGACCTGGAAGAGCAGATCACACCTGTCAGGGCCTCTGATACTGAGC
1 1224137 T C 50 0 0 26 M TCCACCAGGCAACGCGAAAA TTCTGTGTGACAGAGGGGGA 137 GCAGGTGGCCGTCAGACTGGGCCCCCAGGACCCCGAACAGCCAGACGGATGCCGCCGGCCCAGGACTCCATGGCTGCGTGAACCTGCCACCAACAGCGACAGGCTCCACCAGGCAACGCGAAAAGCGTCCGGGACGTGGGGCTTGTGGGG[C/T]GCCGGGCCACGCCAGAGACACTGGGGAAGGTGGATGCCACGGTCCCCTCCCCGGGGAAACACATGGACCCTCCCCCTCTGTCACACAGAATCACCACCAGACCCCACTCTCCAACCACAGCTTCCAAAAGTCACACTCTTCACGTTTTA
1 1224160 G A 50 0 7 14 H TCCACCAGGCAACGCGAAAA TTCTGTGTGACAGAGGGGGA 137 CCCAGGACCCCGAACAGCCAGACGGATGCCGCCGGCCCAGGACTCCATGGCTGCGTGAACCTGCCACCAACAGCGACAGGCTCCACCAGGCAACGCGAAAAGCGTCCGGGACGTGGGGCTTGTGGGGTGCCGGGCCACGCCAGAGACACT[A/G]GGGAAGGTGGATGCCACGGTCCCCTCCCCGGGGAAACACATGGACCCTCCCCCTCTGTCACACAGAATCACCACCAGACCCCACTCTCCAACCACAGCTTCCAAAAGTCACACTCTTCACGTTTTATTTTATTTTTTGAGATGGAGTCT
1 1224783 G A 50 0 19 16 H GGAGACCCCACCTCTACAAA GTAGCTGAAACCACAGGTGC 74 AGGACGCAGTGGCCACACCTATAATCCCAGCCCTTTGGGAGGCTAAGACAGGTGGATCACTTAAGCCCAGAAGTTCAAGACCAGCTTGGGCAACATAGGGAGACCCCACCTCTACAAAAACAATTACGAAAATTCGCCAGGCGCGGTGGC[A/G]TGCACCTGTGGTTTCAGCTACTCAAGAGGCTGAGGTGGGAGGCCTCCCGAACCTGGAATGCGAAGGTTGCAGTGAACTGTGATCACACCACTGCACTCCAGCCTGGGCGGCATACCGAGACCCTGACTCAAAACAAGACAAAACAAAAT
左から、
ターゲット(ERR194146)では、26リードが変異型で検出されたことがわかります。hg38から作成したコントロール配列では、リファレンス型が50リードで変異型は検出されていません。
に出力されます。
1 36936824 1 36936812 f insertion 1 50 0 33 15 H TTTTTTTTTT GCATGCTAGGGGCTGTTAAG CCGCACCTGTTGTTGTATCC 181 GAATGTCAGACTTTAACCTC TGATGGAACATTGGGATGAG
1 36939873 1 36939870 f insertion 1 50 0 1 36 M C ATCAGAGGGGGACCCTTGTT AGATGACTGTGTCAGGAGGG 73 AGGCCTGGTCCACTACCACT TTGTCCCTGGGCCCTCCTGA
1 36957834 1 36958209 f deletion -397 50 0 7 5 H TGTGTCCCGTGAGCCTGTGTG GAACCTATGTGTCCCGTGAG AGTCATGGGACACACAGGCT 79 GTGTCCCGTGAGCCTGTGTG TGTGCCCCGTGAGCCTGTGT
1 36959937 1 36959973 f deletion -47 50 0 0 13 M TGCCCCATGA ATTCTGTGCCCCATGAGCCT TATGGGGCACACAGACTCAC 85 TGAGCCTGTGTGCCCCATGA TGCCCCATGAGCCTCTGTGC
1 36962666 1 36962642 f deletion -2 50 0 8 12 H AGAGAGAGAGAGAGAGAGAGAGAGA GTGAGAGAGAGGGAGAGGAA CCCTCTCTTTGCCTCTCCAT 84 GTGAGAGAGAGGGAGAGGAA AACAGAAGTGAACAAAAAGT
1 36968265 1 36968222 f deletion -2 50 0 11 7 H GTGTGTGTGTGTGTGTGTGTGTGTGTGTGTGTGTGTGTGTGTGT CCTCACAGTTTCACCTGCTC AGGGAGCATGAACAGAGGGA 106 TCTTTCACTCTCTCTCTCCG TCTTCTTTGTTTCCCTCTGT
1 36986463 1 36986416 f insertion 4 50 0 7 9 H TCCATCCATCCATCCATCCATCCATCCATCCATCCATCCATC CCATCCATCCATCCATCCTG TGGATGGATGGATGGATGGG 78 CATCCTGTCCCCATCTCTCT GCCCATCCATCCATCCATCC
1 37001890 1 37001895 f deletion -9 50 0 25 25 H GGA AGCTCAGTCTTACCACTGGG TCACAACCCCAGCTTCCTCT 70 CTCAGTCTTACCACTGGGGA GGAGACAATGTGGAACTGCC
左からタブ区切りで、
となります。
サポートしているリファレンスゲノム
ref=nameのように表示されたゲノムの名前を指定すると、配列公開サイトから自動的にダウンロードして、データセットが生成されます。
一度作成されると次回以降の実行では、作成されたデータセットをもとに解析が進みます。
Name Description
97103 Water melon (Citrullus lanatus subsp. vulgaris) cv. 97213v2
Asagao1.2 Asagao (Ipomoea nil) Japanese morning glory
B73v4 Corn (Zea mays B73) RefGen v4
Bomo Silkworm (Bombyx mori) Genome assembly (Nov.2016)
Bsubtilis Bacillus subtilis subsp. subtilis str. 168 (NC_000913.3)
Camarosa1.0 Strawberry (Fragaria x ananassa) Camarosa genome assembly v1.0
Camellia20200506 Black tea (Cammellia sinensis) assembly
CharlestonGray2 Water melon (Citrullus lanatus subsp. vulgaris) cv. Charleston Gray v2
Ecoli Escherichia coli str. K-12 substr. MG1655 (NC_000913.3)
GRCm38 Mouse (Mus musculus) Genome Reference Consortium Mouse Build 38
Gifu1.2 Lotus japonicus Gifu
Gmax275v2.0 Soybean (Glycine max) genome project assemble version 2
HBV Hepatitis B virus (strain ayw, NC_003977.2)
IBSC2 Barley (Hordeum vulgare L. cv. Molex) Release 47
IRGSP1.0 Rice (Olyza sativa L. cv. Nipponbare) version 1.0
IWGSC1.0 Wheat (Triticum aestivum L. cv. Chinese Spring) Version 1.0
KOD1 Thermococcus kodakarensis KOD1 (NC_006624.1)
LJ3 Lotus japonicus MG20 v3.0 (Download from https://lotus.au.dk/data/download into LJ3 directory)
Lactuca_sativa Lettuce (Lactuca sativa)
RIB40 Aspergillus oryzae RIB40 (ASM18445v3)
Reikou2.3 Strawberry Reikou genome v2.3
SARS-CoV-2 Severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2, NC_045512.2)
SL3 Tomato (Solanum lycopersicum cv. Heinz 1706) Build 3.0
SScrofa11.1 Pig (Sus scrofa) Release-97
TAIR10 Arabidopsis thaliana version TAIR10
UMD3.1 Cow (Bos taurus L1 Dominette 01449) UMD 3.1
Vcholerae Vibrio cholerae O1 biovar El Tor str. N16961
WBcel235 Caenorhabditis elegans WBcel235
danRer11 Zebrafish (Danio rerio) Genome Reference Consortium Zebrafish Build 11
dmel626 Drosophila melanogaster
hg19 Human (Homo sapiens) Genome Reference Consortium Human Build 19
hg38 Human (Homo sapiens) Genome Reference Consortium Human Build 38
sacCer3 Saccharomyces cerevisiae (UCSC sacCer3)
で任意のリファレンスのデータセットが生成されて解析が進みます。control=controlは省略可能です。
Dockerを用いた解析
Dockerを用いると、dockerhubに登録済みの実行環境をダウンロードして一連の解析を行うことができます。解析が完了すると実行環境はクリアされて結果のみが残ります。
手動でfastq-dumpをインストールして、設定がうまくいかずダウンロードできないような場合は、動作確認済みのDockerの系をお勧めします。
CentOSやUbuntuにインストールする場合
sudo sh get-docker.sh
を実行します。
Ubuntuの場合は、
sudo apt install docker
sudo apt install docker.io
の2つの操作のみでもインストールできます。
でコンテナをダウンロードすることができます。
sudo docker stats
で、止めたいコンテナのCONTAINER IDを調べて、
sudo docker kill CONTAINER_ID
を実行します。
sudo usermod -a -G docker username
を実行(usernameは対象となるユーザのログイン時のIDになります)して、一旦ログアウトして、ログインしなおすとユーザ権限で実行できるようになります。
解析事例
cd ped
perl download.pl accession=SRR11542243
perl ped.pl target=SRR11542243,ref=SARS-CoV-2,clipping=100
miyao:~/ped/SRR11542243$ egrep 'H|M' SRR11542243.bi.snp
1 6312 C A 50 0 0 68 M
1 6312 C G 50 0 0 68 M
1 11083 G T 50 0 0 15 M
1 11157 A T 50 0 69 37 H
1 13730 C T 50 0 0 62 M
1 19524 C T 50 0 1 68 M
1 23929 C T 50 0 1 72 M
1 26576 A T 50 0 62 30 H
miyao:~/ped/SRR11542243$ egrep 'H|M' SRR11542243.sv
1 1208 1 1206 f deletion -1 50 0 92 43 H AA
1 6049 1 6052 f deletion -7 50 0 92 50 H TTT
1 6176 1 6176 f deletion -1 50 0 87 41 H _
1 6701 1 6696 f insertion 1 50 0 89 50 H TTT
1 6781 1 6777 f insertion 1 50 0 86 41 H TT
1 9860 1 9641 r inversion N 50 0 19 13 H ATGTGCTA
1 11083 1 11075 f deletion -1 50 0 0 48 M TTTTTTTT
1 11185 1 11179 f insertion 1 50 0 88 38 H TTTT
1 11222 1 11221 f deletion -1 50 0 90 54 H G
1 11297 1 11292 f insertion 1 50 0 87 38 H TTT
1 12114 1 12111 f insertion 1 50 0 96 51 H T
1 12135 1 12134 f deletion -2 50 0 96 53 H TT
1 13081 1 13078 f deletion -1 50 0 85 44 H TTT
1 15664 1 15660 f insertion 1 50 0 90 54 H TT
1 15677 1 15672 f insertion 1 50 0 92 50 H TTT
1 16094 1 16093 f deletion -1 50 0 86 38 H T
1 16190 1 15688 f insertion 502 50 0 36 31 H G
1 17360 1 17364 f deletion -5 50 0 88 40 H _
1 18692 1 18687 f insertion 1 50 0 90 46 H TTT
1 20913 1 20908 f insertion 1 50 0 85 47 H TTT
1 21950 1 21946 f insertion 1 50 0 87 42 H AA
1 22437 1 21967 f insertion 467 50 0 20 41 H TG
1 23561 1 23580 f deletion -22 50 0 95 43 H TT
1 23561 1 23583 f deletion -23 50 0 95 55 H _
1 23571 1 23582 f deletion -12 50 0 94 50 H _
1 23584 1 23595 f deletion -14 50 0 94 42 H TA
1 24378 1 24374 f insertion 1 50 0 83 45 H TT
1 24953 1 24954 f deletion -2 50 0 95 61 H _
1 25404 1 25402 f deletion -1 50 0 94 43 H TT
1 26660 1 26654 f insertion 1 50 0 97 47 H TTTT
1 26784 1 26816 f deletion -37 50 0 97 47 H TCTT
1 26851 1 26629 f insertion 221 50 0 57 45 H _
1 27216 1 27215 f deletion -1 50 0 68 32 H T
1 27226 1 27225 f deletion -1 50 0 75 41 H G
1 27297 1 27297 f deletion -1 50 0 95 51 H _
1 27444 1 27444 f deletion -2 50 0 87 40 H T
1 29016 1 28340 f insertion 675 50 0 32 17 H _
1 29584 1 29581 f deletion -1 50 0 79 39 H TTT
1 29703 1 29701 f deletion -1 50 0 78 42 H GG
変異の確認例
miyao:~/ped$ perl search.pl SRR11542243 1 26784
# 1 26784 1 26816 f deletion -37
TGGATCACCGGTGGAATTGC GCGCGTACGCGTTCCATGTG 15
Chr1 26784
|
GTTTACAGAATAAATTGGATCACCGGTGGAATTGCTATCGCAATGGCTTGTCTTGTAGGCTTGATGTGGCTCAGCTACTTCATTGCTTCTTTCAGACTGT
|||||||||||||||||||||||||||||||||||||||||||||||||||||| || || || | | || || | || | | |
GTTTACAGAATAAATTGGATCACCGGTGGAATTGCTATCGCAATGGCTTGTCTTTCAGACTGTTTGCGCGTACGCGTTCCATGTGGTCATTCAATCCAGA
| | | | | | | | | | ||||||||||||||||||||||||||||||||||||||||||||||||||
TCGCAATGGCTTGTCTTGTAGGCTTGATGTGGCTCAGCTACTTCATTGCTTCTTTCAGACTGTTTGCGCGTACGCGTTCCATGTGGTCATTCAATCCAGA
|
Chr1 26816
コロナウイルスの配列内に非常によく似た配列があり、塩基置換変異により見かけ上欠失が起こったように見える場合もあるので
注意深く検証する必要があります。
perl download.pl accession=SRR11542244
perl ped.pl target=SRR11542244,ref=SARS-CoV-2,clipping=100
miyao:~/ped/SRR11542244$ egrep 'H|M' SRR11542244.bi.snp
1 9507 A T 50 0 17 13 H
1 11104 T C 50 0 0 72 M
1 12781 C T 50 0 48 37 H
1 19255 C T 50 0 24 18 H
1 24953 G A 50 0 71 73 H
miyao:~/ped/SRR11542244$ egrep 'M|H' SRR11542244.sv
1 4155 1 4756 r inversion N 50 0 28 29 H GGTGTTTTAA
1 5273 1 5271 f deletion -1 50 0 18 16 H GG
1 5872 1 5870 f deletion -1 50 0 68 45 H AA
1 9696 1 9698 f deletion -3 50 0 59 53 H _
1 9860 1 9641 r inversion N 50 0 0 10 M ATGTGCTA
1 11620 1 11616 f insertion 1 50 0 53 27 H TT
RNA-Seqのfastqの配列データは鎖長が一定ではありません。
PEDは一定の鎖長のデータが必要となるため、適当な長さに切りそろえる必要があります。
鎖長の分布は、
perl check_length.pl target=SRR11542243
で確認することができます。
SRR11542243は最大長が101塩基ですが、101塩基で指定するとサンプル数が少なくなるので、
clipping=100で解析しています。この場合は、100塩基以上のリードが選ばれて、100塩基に切りそろえられてから解析されます。
上記の2例は、マレーシアの患者から得られたウイルスですが、塩基置換以外に、挿入・欠失・逆位の変異も入っていることがわかります。
また、100%変異型ではなく半分程度の変異が多く見られることから、感染後に個体内で変異を起こしながら増殖していることがわかります。
また、おなじマレーシア国内で広がったコロナウイルスでも変異が独立していることから、非常に変異を起こしやすいウイルスであることがわかります。
日本晴でバッククロスした個体の自殖個体の早生型と晩稲型の個体のバルクからのNGSデータを解析した。
cd ped
mkdir early
mkdir early/read
mkdir late
mkdir late/read
mv DRR056225_1.fastq DRR056225_2.fastq DRR056226_1.fastq DRR056226_2.fastq early/read
mv DRR056227_1.fastq DRR056227_2.fastq DRR056228_1.fastq DRR056228_2.fastq DRR056229_1.fastq DRR056229_2.fastq DRR056230_1.fastq DRR056230_2.fastq late/read
perl ped.pl target=early,control=late,ref=IRGSP1.0
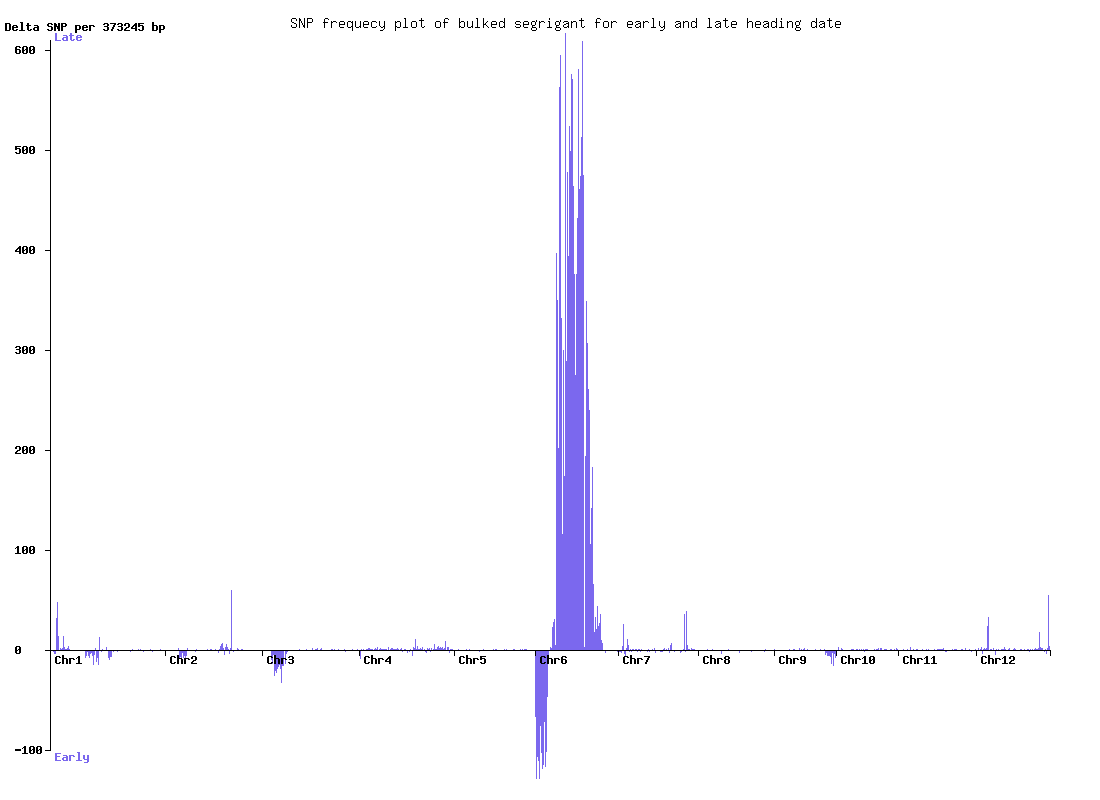
この領域にそれぞれ早生、晩稲に関与する遺伝子座が存在する。
PEDに関する資料
その他のお役立ち情報
現在取扱いをしている企業について
ご自分で解析が困難な場合は、このソフトウェアを用いた解析を受託会社に依頼することもできます。
研究目的での利用はご自由に利用してもらって大丈夫ですが、企業の方で営利目的の場合は、農研機構知的財産部特許ライセンスチーム (電話 029-838-6465 メール naro-kyodaku@naro.affrc.go.jp)にご連絡ください。