Ubuntuのインストール
- Ubuntu Desktop 日本語 Remixより日本語版のISOイメージのファイルをダウンロードします。
- Ubuntu 20.04 LTSインストールガイド【スクリーンショットつき解説】が詳しいので参考にしてインストールしてください。
PEDは解析時間をできるだけ短くするため、CPUの限界近くまで負荷がかかりますのでほかのOSとの共存は避けた方がいいです。
使わなくなった古いコンピュータなどにインストールして専用にするのがお勧めです。
マルチスレッドで動作しますので、4あるいは8コアのCPUを用いると早く処理できます。
PEDのセットアップと解析例
- 必要なソフトをダウンロードとインストールを行います。
$ sudo apt update $ sudo apt upgrade $ sudo apt install curl $ sudo apt install git $ sudo apt install sra-toolkit $ git clone https://github.com/akiomiyao/ped.git
- Ubuntuのバージョンによっては、sra-toolkitのインストールに失敗することがあります。
また最近のバージョンのfastq-dumpは使用前にvdb-configコマンドで実行環境を設定する必要があります。
設定方法は、[fastq-dumpの設定]のページに記載しました。 - NCBIのSRAアーカイブからMiyakojima MG-20のfastq形式のファイル(DRR066832)をダウンロードします。
$ cd ped $ git pull $ perl download.pl accession=DRR066832
git pullで最新のスクリプトに更新されます。git pullは毎回行う必要はありませんが、プログラムが更新されている場合はpullしてください。
pedのディレクトリの中に、DRR066832の名称のディレクトリができて、その中のreadというディレクトリの中にfastqファイルがダウンロードされます。
アメリカからのダウンロードなので時間がかかります。
時々エラーが表示されますが自動的に再接続されるので、気長に待ちます。
同様の構造の任意の名前のディレクトリを作れば手持ちのデータの解析ができます。
- DRR066832の配列をミヤコグサの参照配列gifu1.2にマップして多型を検出します。
$ perl ped.pl target=DRR066832,ref=Gifu1.2
ミヤコグサの参照配列は設定済みなので自動的にダウンロードして解析用データを作成して一連の作業が自動的に進みます。
解析が無事終わるとDRR066832のディレクトリの中に、DRR066832.vcfという結果をまとめたファイルができます。
各座位に対してプライマーが作成可能な場合は、プライマーペアの配列データも出力されます。
Dockerでの解析
- Dockerを用いれば、必要な時に設定済み実行環境を取り込んで解析できます。
グーグルクラウド(GCP)やアマゾンウェブサービス(AWS)などのクラウドでの実行も可能です。 - Dockerのセットアップ
$ sudo apt install docker
$ sudo apt install docker.io
- 解析例
$ sudo docker pull akiomiyao/ped
$ sudo docker run -w /ped -v `pwd`:/work akiomiyao/ped perl download.pl accession=DRR066832,wd=/work
$ sudo docker run -w /ped -v `pwd`:/work akiomiyao/ped perl ped.pl target=DRR066832,ref=Gifu1.2,wd=/work
上記のコマンドをコピーしてターミナルウインドウにペーストして実行してください。
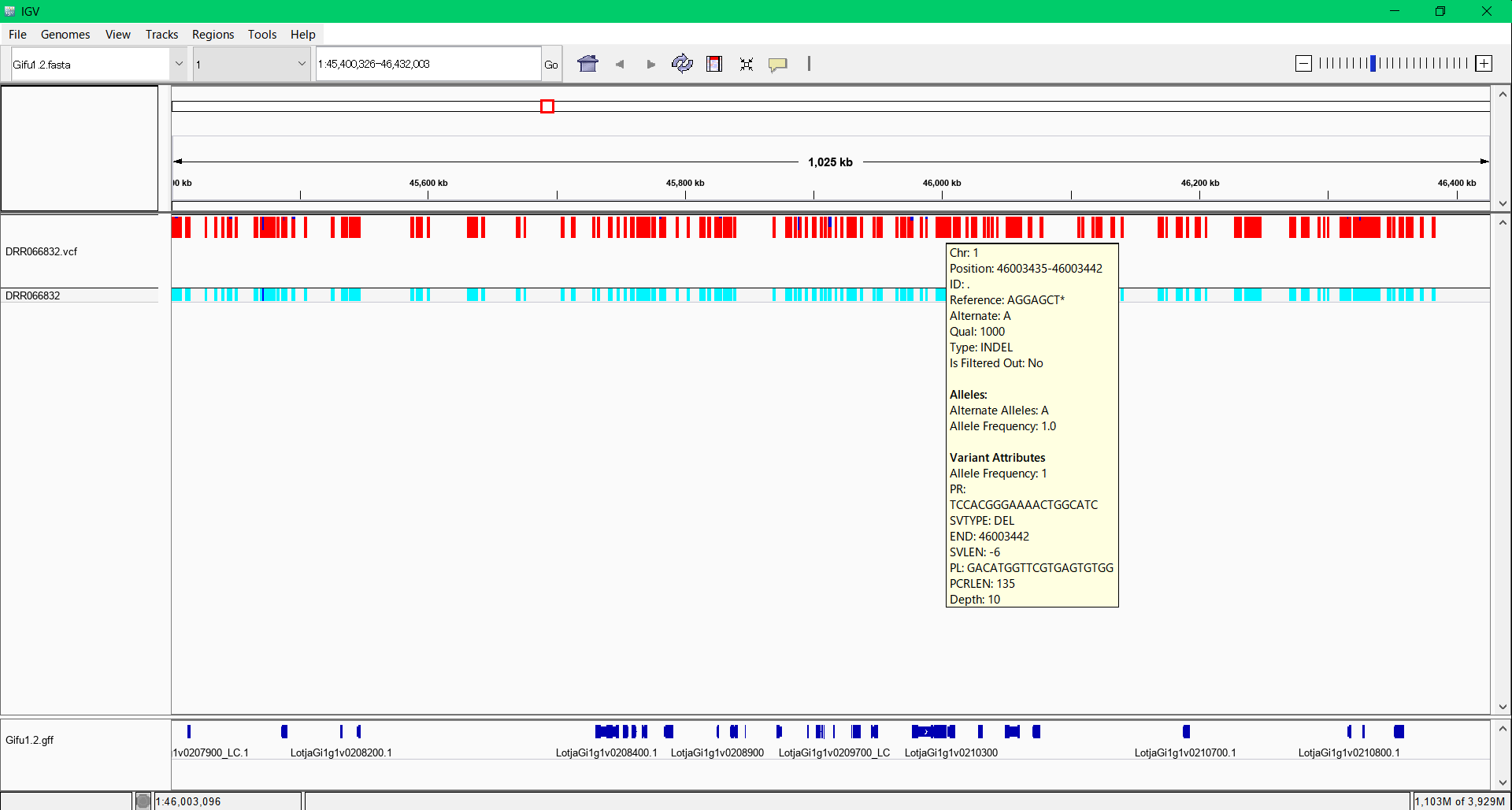
IVGブラウザによる結果の確認
- Integrative Genomics Viewer (IVG) はBroad Instituteで作成されたゲノムブラウザです。
http://software.broadinstitute.org/software/igv/download からダウンロードできます。
Gifu1.2.fastaをクリックしてダウンロードします。
IVGを開いてGenomesのプルダウンメニューよりLoad Genome from Fileを選んで、gifu1.2.fastaをロードします。
Gifu1.2.gff をクリックしてダウンロードして、メインの表示ボックスの下のサブのボックスにドラッグします。
DRR066832.vcf(クリックしてダウンロードできます)
をメインの表示ボックスにドラッグすれば、Miyakojima MG-20の多型データが取り込まれます。
各染色体の番号をクリックすると、染色体ごとの多型情報が表示されます。
興味のある部分をクリックすると、どんどん拡大されます。
多型をマウスで指すと詳細情報が表示されます。
多型の種類や多型頻度、また、プライマー対の配列情報と増幅産物のサイズも表示されます。
ページのトップの画像が表示例です。
多型の部分を増幅するプライマー配列も表示されます。至適配列が見つからない部位では表示されません。
解析事例
- A NIN-LIKE PROTEIN mediates nitrate-induced control of root nodule symbiosis in Lotus japonicus | Nature Communications
ミヤコグサは、マメ科の植物で、根粒菌の共生に関する遺伝子の解析が進められています。根粒菌との共生は土中の窒素源が多い場合は抑制されますが、窒素源の量に関わらず共生が行われる変異体から原因遺伝子を特定した論文です。 - Identification of the causative gene of Lotus japonicus nitrate unresponsive symbiosis 1 mutants
に、コントロールとして野生型のMG-20の配列と、変異体のnrsym1-1とnrsym1-2のNGS配列が公開されています。
- コントロールにMG-20が用いられているので、リファレンスゲノム配列には、GifuではなくMG-20(LJ3)を使用します。
https://lotus.au.dk/data/downloadより、Lotus japonicus MG20 v3.0 GenomeにあるLotusjaponicus_MG20_v3.0_genome.fa.gzをダウンロードします。$ cd ped $ mkdir LJ3 $ mv Lotusjaponicus_MG20_v3.0_genome.fa.gz LJ3 $ cd LJ3 $ gzip -d Lotusjaponicus_MG20_v3.0_genome.fa.gz
として、圧縮状態のgzファイルをfastaファイルに変換します。
また、同時にMG-20のgffファイル Lotusjaponicus_MG20_v3.0_annotations.gff3.gz もダウンロードしておきます。 - ショートリード配列をダウンロードします。DDBJのDRAに登録されているので、https://ddbj.nig.ac.jp/DRASearch/を開いて、
DRR097247 DRR0972478 DRR097249 DRR097250 DRR097251 DRR097252
を1つずつ順にAccessionにペーストしてSearchをクリックします。
検索結果のページの2行目のFASTQのリンクをクリックすると圧縮されたショートリード配列のペアがダウンロードできます。
$ mkdir MG-20 $ mkdir MG-20/read $ mkdir nrsym1-1 $ mkdir nrsym1-1/read $ mkdir nrsym1-2 $ mkdir nrsym1-2/read
として、それぞれコントロールとターゲットのディレクトリを作ります。
$ mv DRR097247_?.fastq.bz2 DRR097248_?.fastq.bz2 MG-20/read $ mv DRR097249_?.fastq.bz2 DRR097250_?.fastq.bz2 nrsym1-1/read $ mv DRR097251_?.fastq.bz2 DRR097252_?.fastq.bz2 nrsym1-2/read
として、ダウンロードしたファイルを、それぞれのディレクトリに移します。 - PEDでの多型検出は、
$ perl ped.pl target=nrsym1-1,control=MG-20,ref=LJ3 $ perl ped.pl target=nrsym1-2,control=MG-20,ref=LJ3
で、それぞれの多型が検出されます。 - LJ3.fasta
LJ3.fasta.fai
Lotusjaponicus_MG20_v3.0_annotations.gff3.gz
を、それぞれダウンロードします。 - 解析済みvcfファイルは、
nrsym1-1.vcf
nrsym1-2.vcf
からダウンロードできます。 - https://igv.org/app/をクリックして、WebのIGVを開きます。
Genomeのプルダウンメニューより、'Local File ...'を選んでダウンロードした、LJ3.fastaとLJ3.fasta.faiを両方一緒に選択してSubmitボタンをクリックします。
Tracksのプルダウンメニューより、'Local File ...'を選んで、Lotusjaponicus_MG20_v3.0_annotations.gff3.gz、nrsym1-1.vcf、nrsym1-2.vcfを順にアップロードします。
第5染色体を選んで、28.1MBのあたりを拡大します。
Lj5g3v1999250.2の遺伝子の中にあるSNPが目的の変異です。
同一遺伝子のコード領域の少しずれた位置に、2つ系統でそれぞれホモ型変異が入っているので、この遺伝子が原因遺伝だろうと見当がつきます。
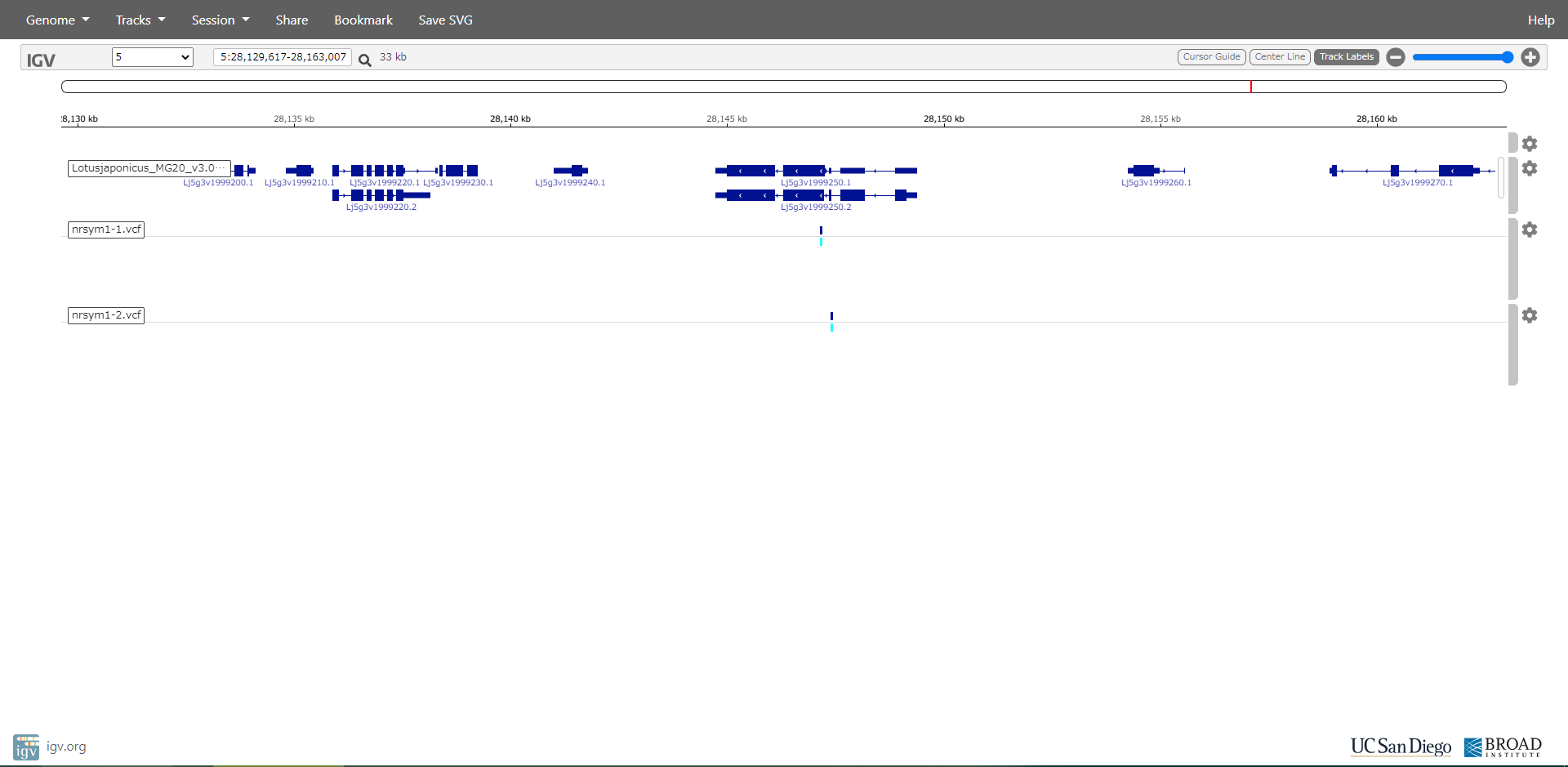
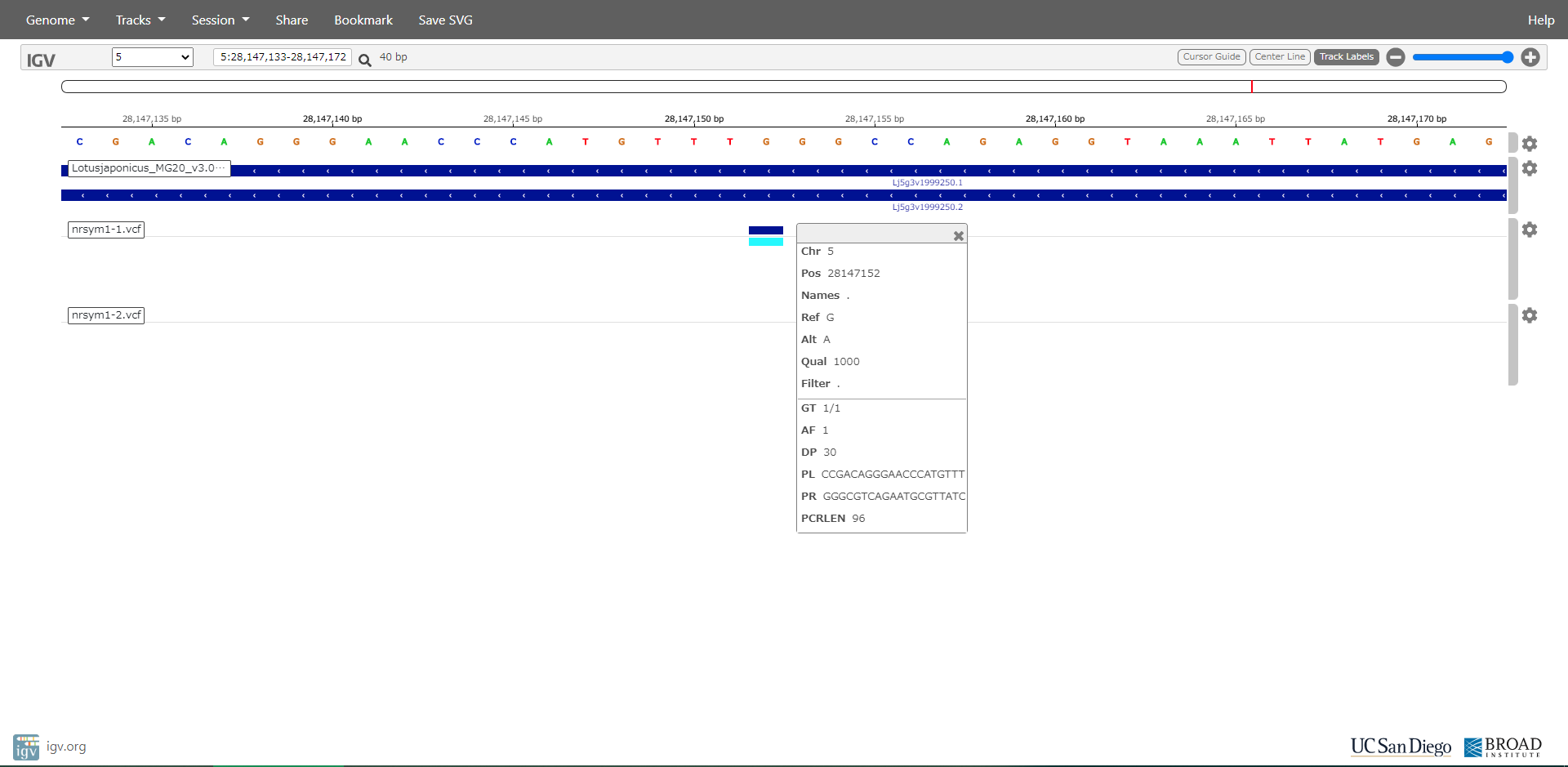
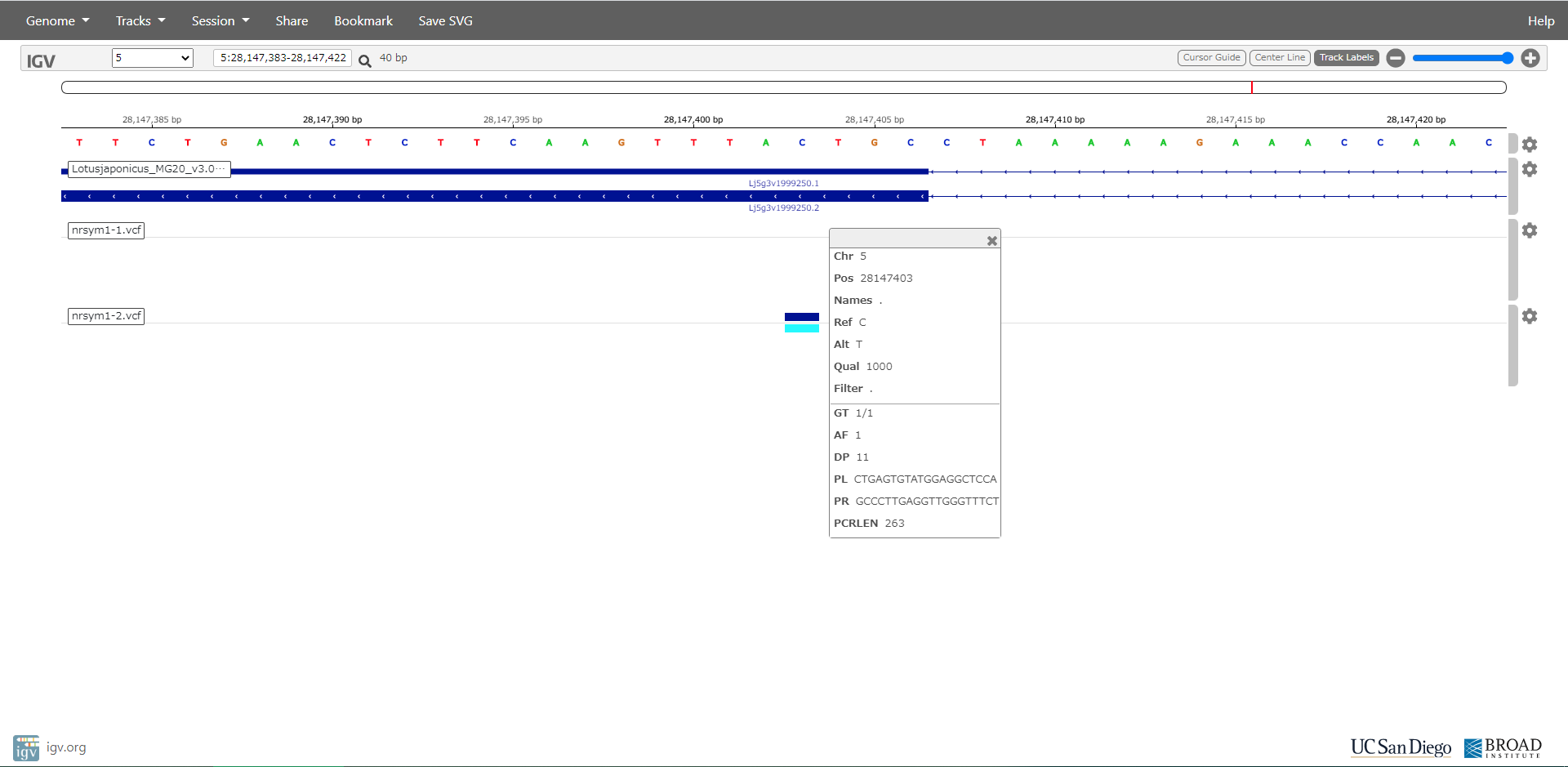
図をクリックすると大きくなります。
論文に掲載されている変異は、nrsym1-2遺伝子の場合で、第5染色体の28,147,403の位置のCからTへの置換変異で、283番目のバリンがイソロイシンに変わっています。
この変異が原因で、窒素源の多少による根粒数の増減の調節ができなくなっていると考えられるので、この遺伝子が窒素源の量による根粒数のコントロールに関与していることが推察されます。 - 注意点 IVGでは長い欠失や転座の情報をうまく表示できない場合があるので、転座と80塩基以上の欠失に関しては、vcfファイルに出力しないように設定しています。
大きな構造変異を調べたい場合は、targetのディレクトリの中に出力された、svの拡張子のついたファイルを直接ご参照ください。
PEDに関する資料
- スクリプト: https://github.com/akiomiyao/ped/
- 論文: Polymorphic edge detection (PED): two efficient methods of polymorphism detection from next-generation sequencing data (2019) BMC Bioinformatics 20(1):362
お問い合わせ
〒305-8518茨城県つくば市観音台2-1-2
農研機構作物研究部門
宮尾安藝雄 (miyao@affrc.go.jp)