IGV Web Application
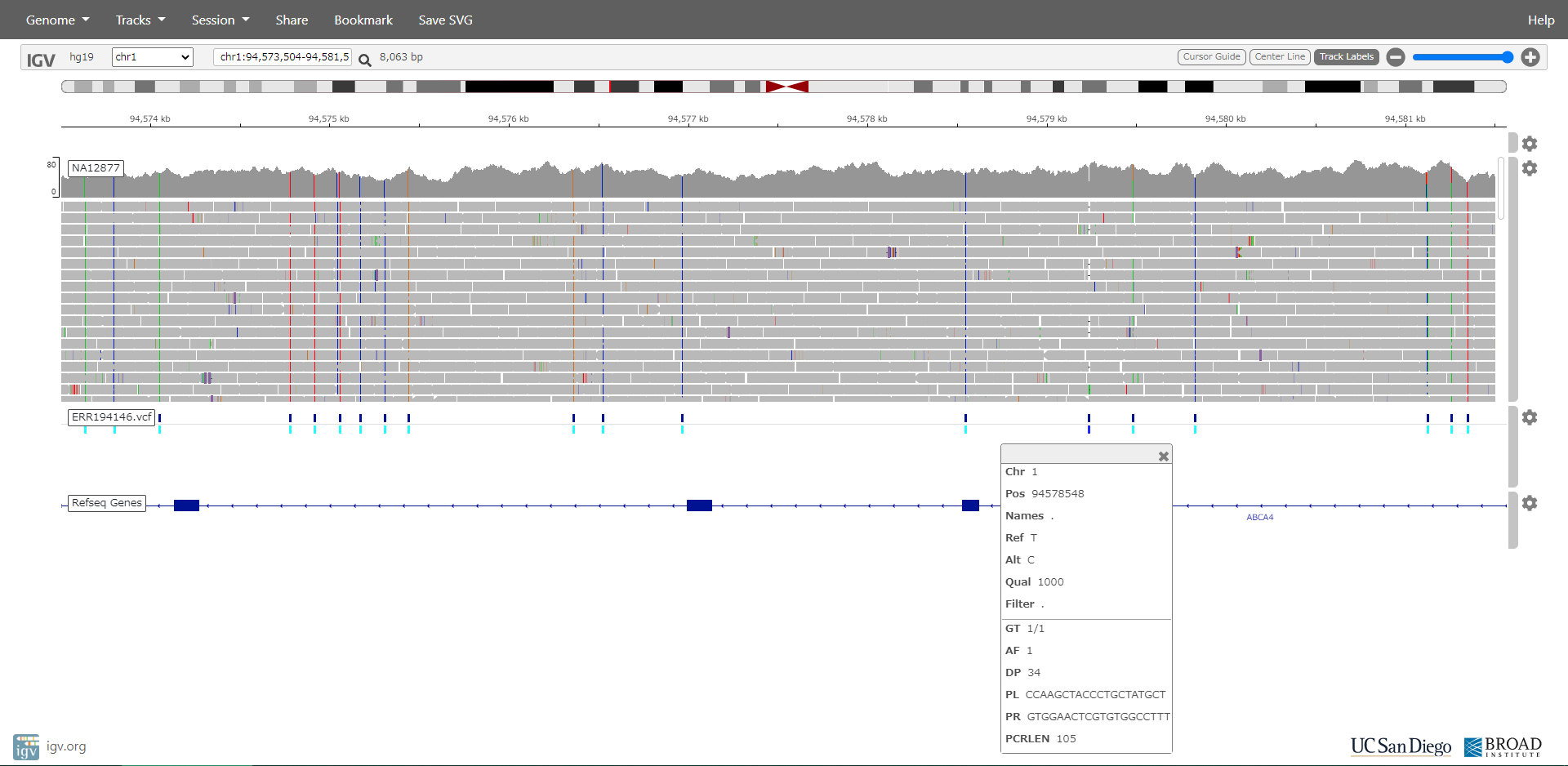
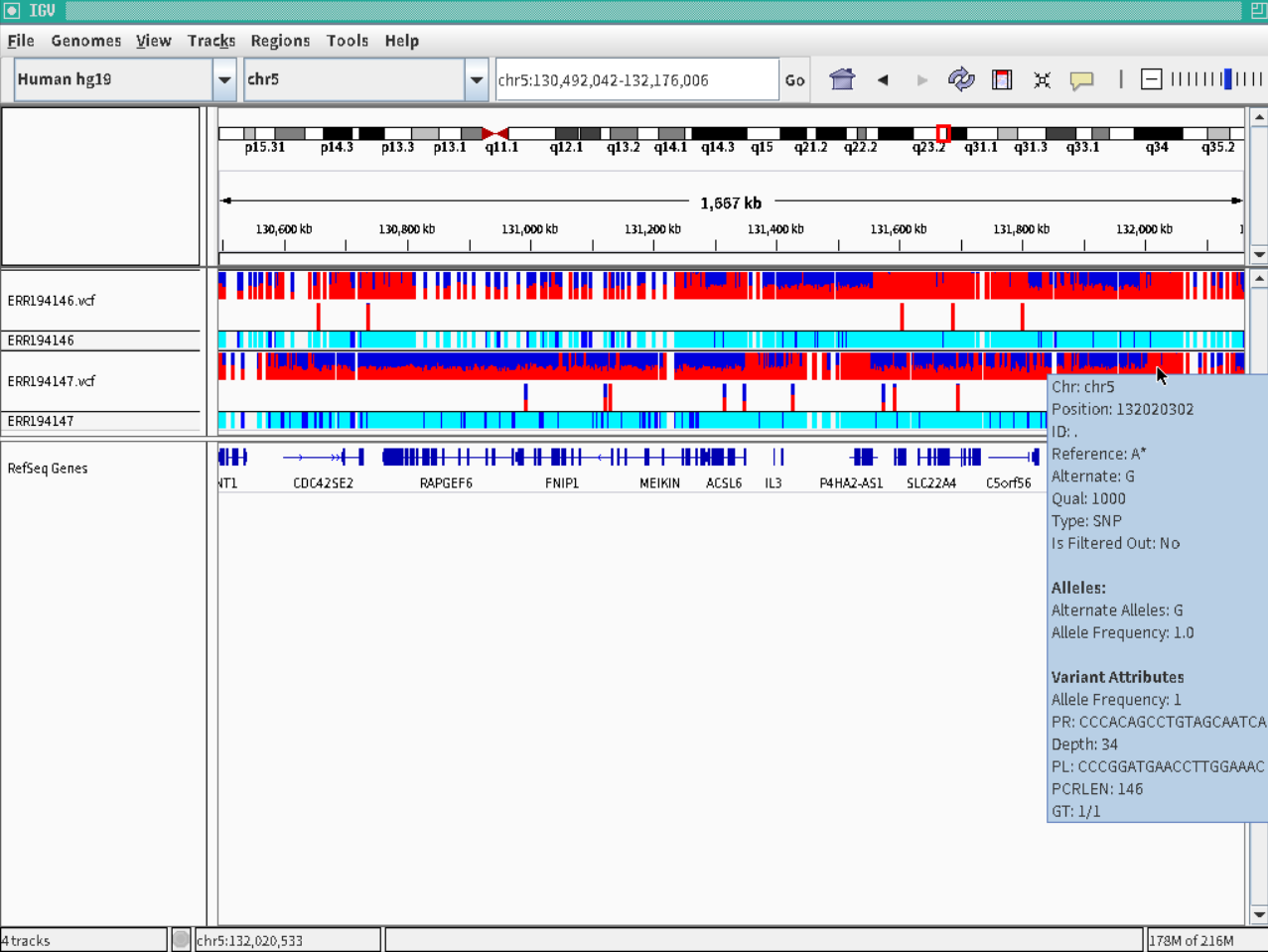
ERR194146.vcf ERR194146.vcf.idx
ERR194147.vcf ERR194147.vcf.idx
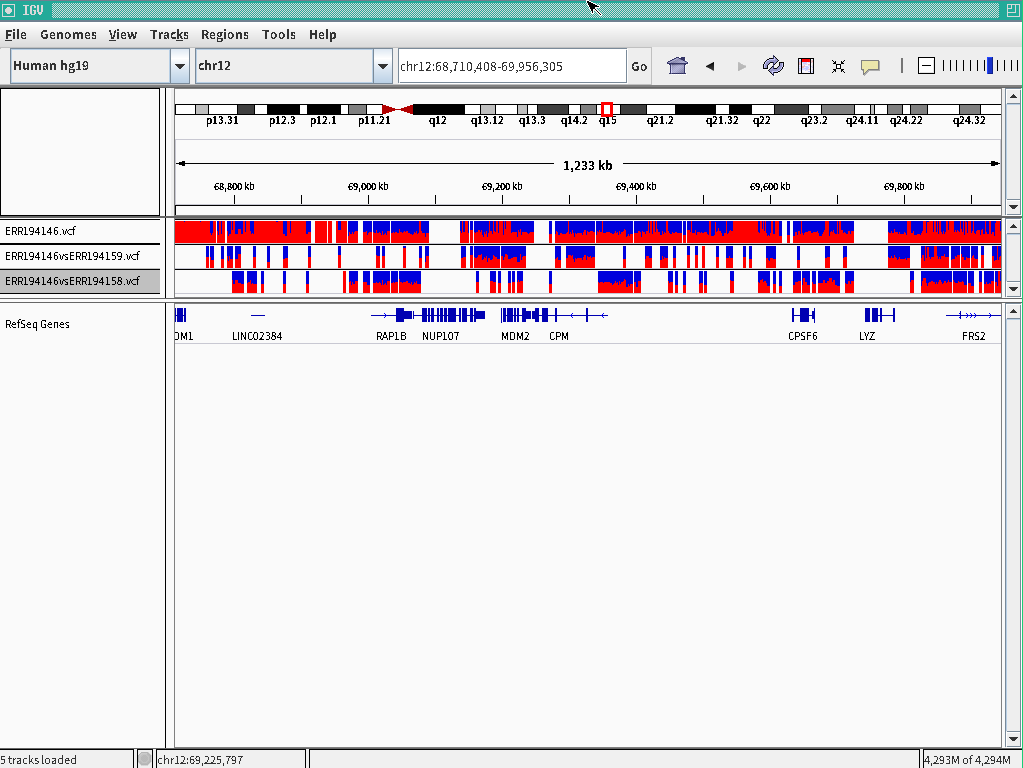
ERR194146vsERR194158.vcf (母親特異的多型のvcfファイル)
ERR194146vsERR194159.vcf (父親特異的多型のvcfファイル)



$ sudo apt update $ sudo apt upgrade $ sudo apt install curl $ sudo apt install git $ sudo apt install sra-toolkit $ git clone https://github.com/akiomiyao/ped.git
$ cd ped $ git pull $ perl download.pl accession=ERR194146 $ perl download.pl accession=ERR194147git pullで最新のスクリプトに更新されます。
$ perl ped.pl target=ERR194146,ref=hg19 $ perl ped.pl target=ERR194147,ref=hg19ヒトの参照配列は設定済みなので自動的にダウンロードして解析用データを作成して一連の作業が自動的に進みます。
$ perl ped.pl target=ERR194146,control=ERR194158,ref=hg19 $ perl ped.pl target=ERR194146,control=ERR194159,ref=hg19コントロール側には存在しない多型のみが出力されます。
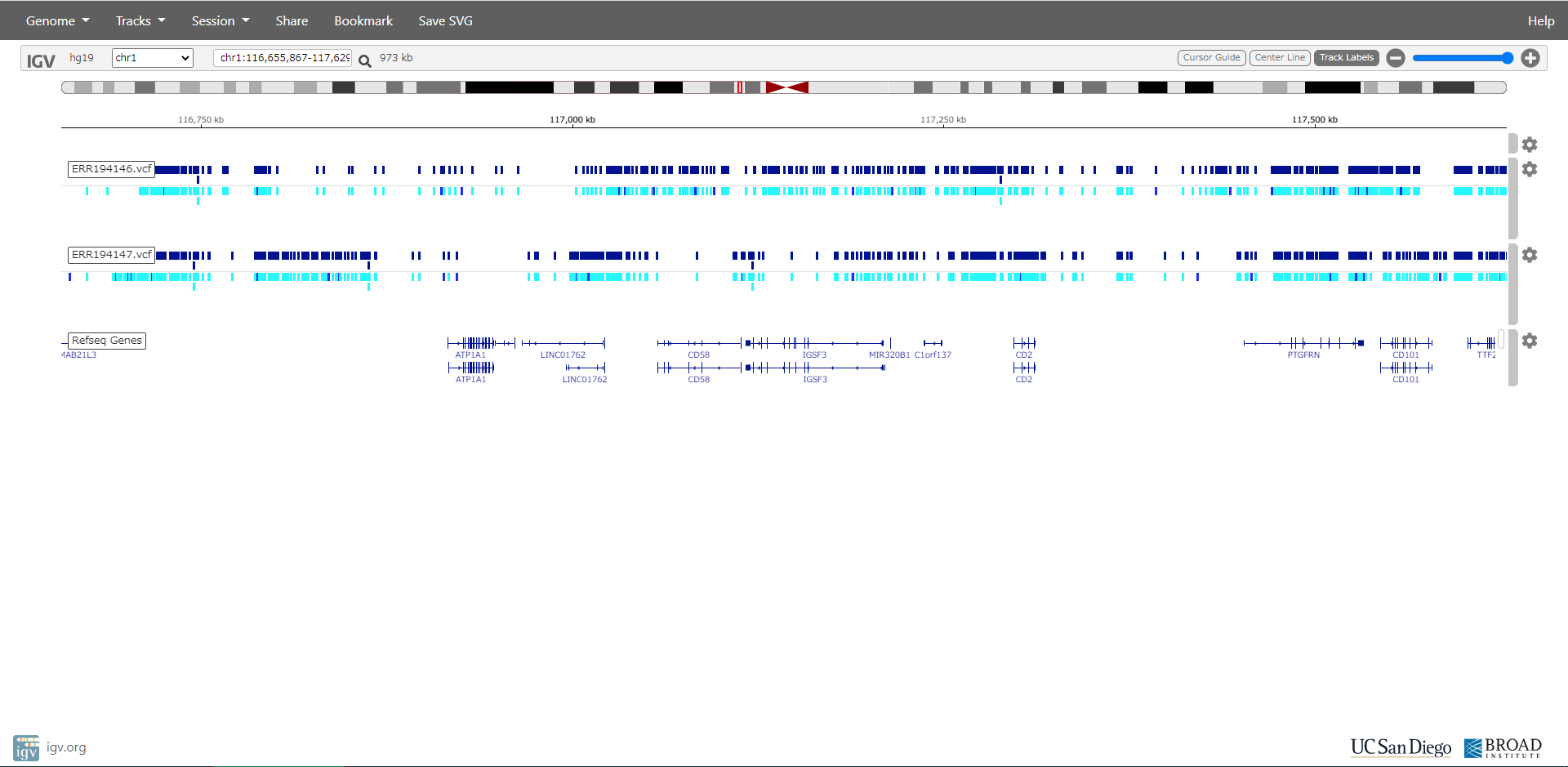
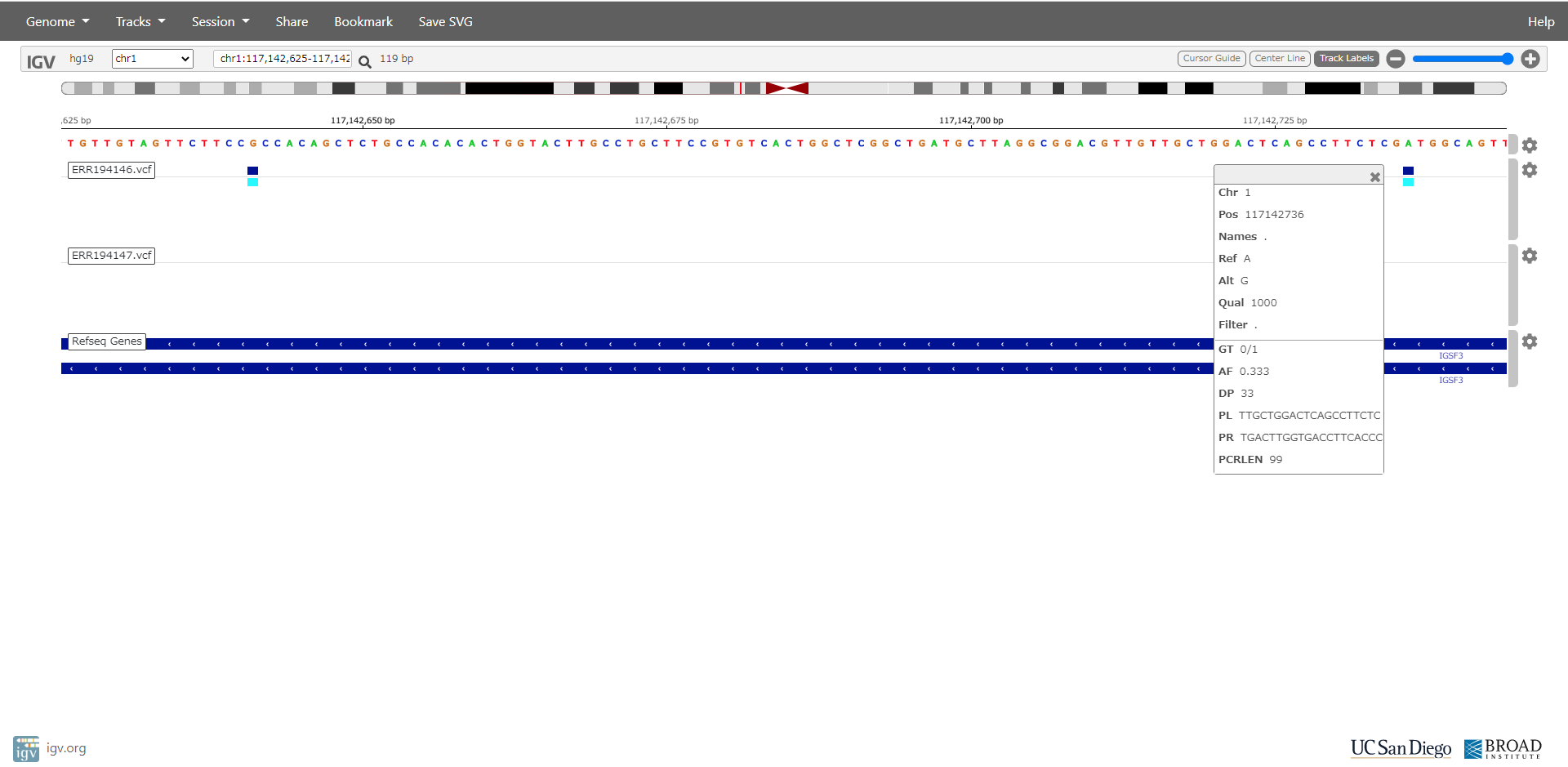
 図をクリックすると大きくなります。
図をクリックすると大きくなります。